論文解説 其の一
論文の情報
Identification of novel avian and mammalian deltaviruses provides new insights into deltavirus evolution.
Iwamoto M, Shibata Y, Kawasaki J, Kojima S, Li YT, Iwami S, Muramatsu M, Wu HL, Wada K, Tomonaga K, Watashi K, Horie M*.
Virus Evol. 2021 Feb 12;7(1):veab003. doi: 10.1093/ve/veab003
オープンアクセス論文なのでどなたでもご覧いただけます。こちらから論文にアクセスできます。
論文解説第一弾ですが、かなりボリュームのある内容なので長文になってしまいました。
論文の解説
デルタウイルスの概要と歴史
デルタウイルスは約1,700塩基の小さなゲノムを持つRNAウイルスです。デルタウイルスは自分だけでは感染性のウイルス粒子を作れません。そのため、ウイルス粒子の形成には他のウイルスのタンパク質を必要とします。そのためデルタウイルスはサテライトウイルスと呼ばれています(サテライトウイルスのウイルス粒子形成を助けるウイルスをヘルパーウイルスと呼びます)。
1970年代にデルタウイルスであるD型肝炎ウイルス(Hepatitis delta virus: HDV)が発見されました。しかし、2018年までHDV以外のデルタウイルスは知られておらず、HDVの起源やその進化はわかっていませんでした。2018年以降、ヘビや鳥、げっ歯類動物や無脊椎動物など、様々な動物から多様なデルタウイルスが発見され、徐々にデルタウイルスの進化が明らかになりつつありました。しかし、これらの多様なデルタウイルスはHDVとは遺伝的に極めて遠く、系統樹上の大きなギャップが存在していたため、 HDVの起源は依然としてわかっていませんでした。
また、HDVのヘルパーウイルスとしてB型肝炎ウイルス(Hepatitis B virus: HBV)が知られています。またヘビのデルタウイルスのヘルパーウイルスとしてアレナウイルスが同定されています。しかし、上記の通り、これらのウイルスは遺伝的に遠いため、どのようにサテライト/ヘルパーの関係が進化してきたのかもわかっていませんでした。
新規デルタウイルスの発見と種間伝播
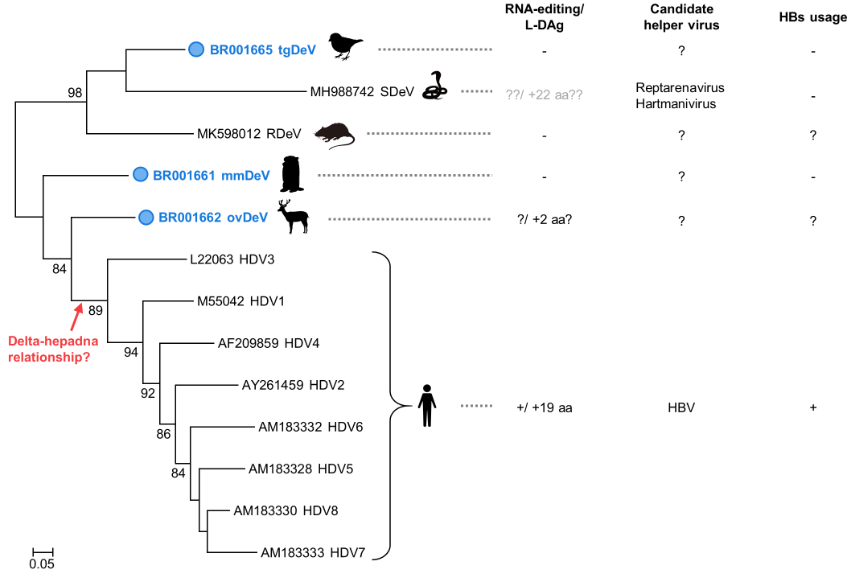
本研究ではデルタウイルスの進化およびデルタウイルスとヘルパーウイルスの共進化を解明するため、新規デルタウイルスの探索を行いました。公共のデータベースの大規模検索(鳥類および哺乳類動物由来の約46,000以上のRNA-seqデータ)を行い、鳥類(キンカチョウ)および哺乳類動物(オジロシカ、ウッドチャック)から計3種の新規デルタウイルスを発見しました。
これらの新規ウイルスの配列を用いて分子系統樹解析を行ったところ、これらの新規デルタウイルスのうち哺乳動物由来の2種のデルタウイルスが、HDVに比較的近縁であり、これまでの系統樹上のギャップを埋めるウイルスであることが示されました。また、HDVは哺乳動物のデルタウイルスを起源とすることが示唆されました。
また、樹形が宿主生物の進化の系統樹とは一致しないことから、デルタウイルスは進化の過程において種間伝播を経てきたことが示唆されました(注1)。さらに鳥由来の検体を用いてデルタウイルスを検出、さらに全ゲノム配列を同定しました。その結果、遺伝的に極めて類似したデルタウイルスが、スズメ目の様々な種の鳥に感染していることがわかり、スズメ目の鳥において、ごく近年にデルタウイルスの種間伝播が起こったことが示唆されました。
デルタウイルスとヘルパーウイルスの共進化
また発見したデルタウイルスについて、ヘルパーウイルスとの共進化に関する解析も行いました。はじめにデルタウイルスのRNA編集によるアイソフォームの発現に着目しました。HDVは宿主のRNA編集機構を利用することによって、1つの遺伝子から2つのアイソフォームのタンパク質を発現します。HDVの場合、ストップコドンのRNA編集により19アミノ酸長いタンパク質が発現します。この長いタンパク質のC末端にはHBVのエンベロープタンパク質と相互作用するための重要な領域が含まれています。
本研究では、公共のRNA-seqデータを用いて、RNA編集が起こっているかどうかを検討しました。その結果、オジロシカのデルタウイルスでのみRNA編集が起こっていることを示唆するデータが得られました。これらのことから、オジロシカのデルタウイルスとヒトのHDVの共通祖先において、宿主のRNA編集機構を利用するという形質が獲得されたことが示唆されました。しかし、オジロシカのデルタウイルスの場合、RNA編集が起こったとしても2アミノ酸しかタンパク質は伸長されず、HBVのエンベロープタンパク質と相互作用するためのモチーフ配列は存在しません。
さらに、実験的な解析も行いました。上記の通りHBVがHDVのヘルパーウイルスとして知られています。そこで、今回発見したデルタウイルスが、HBVのエンベロープタンパク質を利用してウイルス粒子を形成できるかどうかを検討しました。本研究では諸事情により鳥とウッドチャックのデルタウイルスしか検討することができませんでしたが、両ウイルスはHBVのエンベロープタンパク質の存在下においても感染性の粒子を形成できないということが示されました。これらのことから、HDV/HBVで見られるヘルパー/サテライトの関係は、動物のデルタウイルスとHDVの分岐以降に成立したと考えられました。
本研究の重要性と今後の展望
本研究においてこれまでの系統樹のギャップを埋めるような新規デルタウイルスを発見し、ウイルスの多様性と進化という観点から重要な知見が得られました。さらに、コンピューター解析や実験的解析によって、サテライト/ヘルパーの関係についても新たな知見が得られました。
一方、これらの新規デルタウイルスのヘルパーウイルスはまだ見つかっていません。デルタウイルスを検出した公共のデータベースにおいて、網羅的なウイルスの検出を試みましたが、エンベロープウイルスの共感染を示す結果は得られませんでした。今後は動物検体等を用いて、ヘルパーウイルスを同定する必要があります。
また、感染症という観点においても重要な知見が得られました。過去にデルタウイルスが種間伝播を起こしてきたことから、ヒトや動物の新興感染症の原因となることも考えられます。ヒトのHDVはHBVと共感染することによって、B型肝炎の症状を悪化させることが知られています。本研究においてデルタウイルスが検出された動物が何らかの疾病を示していたのかどうかはわかりませんでした。今後は疫学的な調査も必要になると思います。
注釈
注1)ウイルスが宿主生物と共分岐してきた場合は、それぞれの樹形が(限りなく)一致します。
論文の裏話?
本研究は、堀江が初めて本格的なコンピューター解析を行った論文です。また「コンピュータ解析で得られた知見をもとに、動物由来の検体からのウイルスの探索や実験ウイルス学によってウイルスの進化につながる知見を得る」という、思い描いていた通りの研究(実際は山あり谷ありでしたが)ができました。
また本研究では数々のすばらしい共同研究を展開することができました。科研費・新学術領域ネオウイルス学の班会議において新規デルタウイルスの発見に関する発表した後に、世界でも稀なデルタウイルスの実験研究者である岩本将士さん(この論文の第一著者でもあります)から声をかけてもらい、新規デルタウイルスの人工合成システムの作成、さらにはヘルパーウイルスについての実験的な検証をすることができました。さらに分子生物学会においても同様の発表をしたところ、感染研の渡士先生からも声をかけていただき、台湾のウッドチャックの研究者との共同研究にも発展しました。
さらには鳥のデルタウイルスについては北大の和多先生に連絡したところ、迅速にご協力いただき、実際の動物検体からデルタウイルスを検出し、ゲノム全長を決めることができました。これにより、鳥のデルタウイルスが近年に種間伝播を経たということが明らかとなりました。
自画自賛になりますが(笑)、本当に良い論文で自信の一作です。また査読者からも大絶賛を受け、「I need to congratulate the authors on an absolutely stellar piece of work.」「Absolutely tour de force.」「By far the best study of its kind undertaken so far.」「this is fantastic study. Hats off.」などのコメントをいただき、励みになりました。



